
研究员 /taoyong.ac@gmail.com/肿瘤表观遗传:细胞命运决定与肿瘤发生发展
2009年于中国科学院遗传与发育生物学研究所获博士学位。2009-2014年任中国科学院基因组所助理研究员,副研究员。2014-2022年先后在约翰霍普金斯大学医学院,福瑞德.哈金森肿瘤肿瘤研究中心从事博士后研究。2022年入职best365官网登录入口任职研究员。
研究方向:细胞命运决定与肿瘤发生发展
陶勇研究组以组织类器官、肿瘤细胞系、肿瘤类器官、小鼠等为模式,运用遗传、表观遗传、基因组学以及细胞生化等研究方法,研究结肠干细胞细胞命运决定和结肠癌发生之间的关系以及雄性激素受体阳性前列腺癌在治疗过程中发生细胞命运转分化的分子机制,并力求增进结肠癌、前列腺癌的临床管理。
1. 发展定点表观遗传编辑工具
癌症研究的一个基本目标就是鉴定所有具有能够驱动肿瘤生长的关键改变,包括DNA突变和表观遗传修饰改变。鉴定表观遗传改变中的驱动性改变因缺乏相应研究方法而处于滞后的状态。我们以系统鉴定出肿瘤细胞特异表观遗传改变中具有重要功能意义的部分为研究目的,计划开发基于慢病毒颗粒的表观遗传编辑系统,并以开发的工具为基础,以结肠癌为研究模型,测试利用定点表观遗传编辑工具鉴定肿瘤细胞特异表观遗传改变中驱动事件。
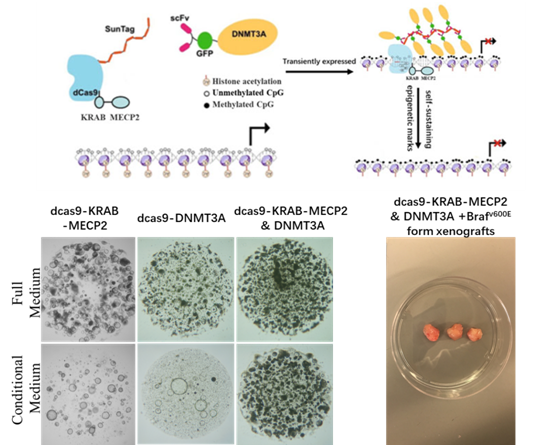
图1.通过定点表观遗传编辑工具沉默特定基因或基因组合,结肠类器官干性增强,分化受阻,同时促进结肠癌的发生。
2. 右端结肠去分化的分子机制
衰老相关的DNA甲基化改变是癌症异常甲基化改变的起源之一,衰老相关的DNA甲基化改变阻止了小鼠右端结肠干细胞分化,并促进结肠肿瘤的产生(Tao el. al., Cancer Cell 2019)。但具体哪些基因受到异常表观修饰影响,同时导致右端结肠干细胞分化受阻仍有待深入地研究。
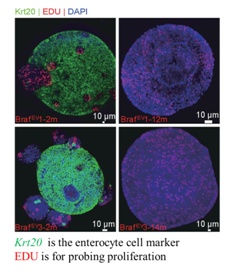
图2. 小鼠右端结肠结肠系统在体外长时间培养后,细胞分化受阻,干性增强。
3. 左、右端结肠癌存在系统差异的机制
左、右半结肠癌存在明显的异质性, 其在组织病理类型、分子特征、流行病学、临床表现上差异明显, 其在治疗、复发、预后上也均存在差异, 左、右半结肠癌这些差异特别是分子特征差异产生的机制仍还不清楚,有待研究。
4. 雄性激素受体阳性前列腺癌发生细胞命运转分化的分子机制
前列腺瘤细胞生长具有雄激素信号通路的依赖性。其原理是雄激素与雄激素受体(AR)结合,作用于前列腺肿瘤细胞,促进前列腺肿瘤细胞的增殖。临床上对于AR阳性前列腺癌患者,采取雄激素撤除或抗雄激素及抗雄激素受体的内分泌治疗往往有效。但是,部分患者在内分泌治疗一到两年后会对治疗不再敏感,转变为激素难治性前列腺癌,其生存期一般不超过12个月,其中有大于20%的去雄抵抗前列腺癌发展为AR阴性前列腺癌,包括双阴性前列腺癌(DNPC)和神经内分泌前列腺癌(NEPC)。然而雄性激素受体阳性前列腺癌发生细胞命运转分化的分子机制仍还不清楚,有待研究。
组员
2021级硕士研究生:杨晗,杨枟翰
代表性论文
1. Zhang Y, Kohrn BF, Yang M, Nachmanson D, Soong, R Lee IH, Tao Y, Clevers H, Swisher WM, Brentnall TA, Loeb LA, Kennedy SR, Salk JJ, Naxevora K, Risques RA Risques (2021). PolyG-DS: An Ultra-Sensitive Polyguanine Tract Profiling Method to Detect Clonal Expansions and Trace Cell Lineage. Proceedings of the National Academy of Sciences, 2023373118. PMID: 34330826
2. Lu Z#, Zou J#, Li S#, Topper M, Tao Y, Zhang H, Xie W, Kong X, Vaz M, Li H, Cai Y, Limin Xia , Rodgers K, Lee B, Riemer J, Jiao X, Yen R, Day C, Zhang W, Cui Y, Easwaran H, Hulbert A, Kim K, Zhou S, Yang S, Battafarano R, Bush E, Broderick S, Cattaneo S, Brahmer J, Rudin C, Wrangle J, Mei Y, J Kim Y, Zhang B, Wang K, Forde P, Margolick J, Nelkin B, Zahnow C, Pardoll D, Housseau F*, Baylin S* , Shen L*, Brock M*. Adjuvant Epigenetic Therapy Inhibits Lung Metastases by Disrupting the Pre-Metastatic Niche. Nature. 2020. PMID: 32103175
3. Kong X #, Chen J#, Xie W#, Brown S, Cai Y, Wu K, Fan D, Nie Y, Tiedemann R, Tao Y, Chiu Yen R, Topper M, Zahnow C, Easwaran H, Rothbart S*, Xia L* and Baylin S*. “Defining UHRF1 Domains That Support Maintenance of Human Colon Cancer DNA Methylation and Oncogenic Properties”. Cancer Cell. 2019 Apr 15; 35(4) 633-648.e7. PMID:30956060.
4. Tao Y#, Kang K#, Petkovich D, Bhandari Y, In J, Stein-O'Brien G, Kong X, Xie W, Zachos N, Maegawa S, Brown S, Chiu Yen R, Shao X, Thakor J, Lu Z, Cai Y, Zhang Y, Gonzalez I, Peinado M, Zahnow C, Ahuja N, Fertig E, Issa J, Baylin S*, Easwaran H*. Aging-like spontaneous epigenetic silencing facilitates Wnt activation, stemness and BrafV600E-induced tumorigenesis. Cancer Cell. 2019 Feb 11;35(2):315-328.e6. PMID: 30753828. (Comment in The Origin of CIMP, At Last. Ushijima T, Suzuki H. Cancer Cell, 2019; Faculty Opinions Recommendations: )
5. Zhang Y#, Li Y#, Li T#, Shen X#, Zhu T, Tao Y, Li X, Wang D, Ma Q, Hu Z, Liu J, Ruan J, Cai J, Wang H*, Lu X*. Genetic Load and Potential Mutational Meltdown in Cancer Cell Populations. Molecular biology and evolution.2019 Jan 15; 36(3): 541-552.
6. Xie W, Kagiampakis I, Pan L, Zhang Y, Murphy L, Tao Y, Kong X, Kang B, Xia L, Carvalho F, Sen S, Chiu Yen R, Zahnow C, Ahuja N, Baylin S*, Easwaran H*. (2018). DNA Methylation Patterns Separate Senescence from Transformation Potential and Indicate Cancer Risk. Cancer cell Feb, 12; 33(2), 309-321 e305. PMID: 29438699.
7. Wang HY, Chen Y, Tong D, Ling S, Hu Z, Tao Y, Lu X, Wu CI. Is the evolution in tumors Darwinian or non-Darwinian? National Science Review 5 (1), 15-17.
8. Tao Y#, Hu Z#, Ling S#, Yeh SH, Zhai W, Chen K, Li C, Wang Y, Wang K, Wang HY, Hungate EA, Onel K, Liu J, Zeng C, Hudson RR, Chen PJ, Lu X*, Wu CI*. Further genetic diversification in multiple tumors and an evolutionary perspective on therapeutics. bioRxiv 2015.
9. Ling S#, Hu Z#, Yang Z#, Yang F#, Li Y, Lin P, Chen K, Dong L, Cao L, Tao Y, Hao L, Chen Q, Gong Q, Wu D, Li W, Zhao W, Tian X, Hao C, Hungate EA, Catenacci DV, Hudson RR, Li WH, Lu X*, Wu CI*. Extremely high genetic diversity in a single tumor points to prevalence of non-Darwinian cell evolution. Proc Natl Acad Sci U S A. 2015 Nov 24;112(47):E6496-505. PMCID: 4664355.
10. Tao Y, Ruan J, Yeh SH, Lu X, Wang Y, Zhai W, Cai J, Ling S, Gong Q, Chong Z, Qu Z, Li Q, Liu J, Yang J, Zheng C, Zeng C, Wang HY, Zhang J, Wang SH, Hao L, Dong L, Li W, Sun M, Zou W, Yu C, Li C, Liu G, Jiang L, Xu J, Huang H, Li C, Mi S, Zhang B, Chen B, Zhao W, Hu S, Zhuang SM, Shen Y, Shi S, Brown C, White KP, Chen DS, Chen PJ, Wu CI. Rapid growth of a hepatocellular carcinoma and the driving mutations revealed by cell-population genetic analysis of whole-genome data. Proc Natl Acad Sci U S A. 2011 Jul 19;108(29):12042-7. PMCID: 3141952.
11. Gan Q, Bai H, Zhao X, Tao Y, Zeng H, Han Y, Song W, Zhu L, Liu G. Transcriptional Characteristics of Xa21‐mediated Defense Responses in Rice. J Integr Plant Biol. 2011 Apr;53(4):300-11. PMID: 21324061.
12. Song Q, Liu G, Hu S, Zhang Y, Tao Y, Han Y, Zeng H, Huang W, Li F, Chen P, Zhu J, Hu C, Zhang S, Li Y, Zhu H, Wu L. Novel autoimmune hepatitis-specific autoantigens identified using protein microarray technology. J Proteome Res. 2010 Jan;9(1):30-9. PMCID: 2801765.
13. Lin YY, Liu C, Chien WH, Wu LL, Tao Y, Wu D, Lu X, Hsieh CH, Chen PJ, Wang HY, Kao JH, Chen DS. New Insights into the Evolutionary Rate of Hepatitis B Virus at Different Biological Scales. J Virol. 2015 Apr;89(7):3512-22. PMCID: 4403390.
本实验室长期招聘博士后和助理研究员。


